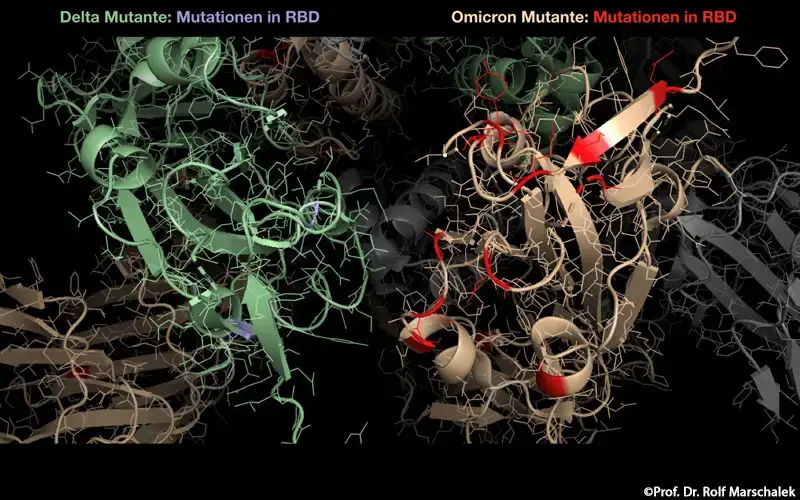
Omikron ist mit einem ungewöhnlich starken Anstieg der COVID-19-Fälle in der südafrikanischen Provinz Gauteng, zu der auch die Großstadt Johannisburg gehört, assoziiert. Die von der Weltgesundheitsorganisation (WHO) als besorgniserregend eingestufte Variante (VOC) wird in immer mehr Ländern registriert. Obwohl noch viele Informationen fehlen, sind Wissenschaftler und Politiker gleichermaßen besorgt. Omikron weist mehr Veränderungen auf als die bisher bekannten Varianten und vereint erstmals kritische Mutationen von Alpha, Beta, Gamma und Delta in der Rezeptorbindungsdomäne (RBD). Diskutiert werden eine höhere Virulenz und leichtere Übertragbarkeit. Befürchtet wird weiterhin, dass Omikron die derzeit verwendeten COVID-19-Impfstoffe unterwandern und einer Erkennung oder Abwehr durch das Immunsystem entgehen könnte (Immunevasion).
Charakteristik
Omikron ist insbesondere wegen der großen Anzahl an Mutationen im Spike-Gen (S-Protein) besorgniserregend. Viele dieser Varianten befinden sich in der RBD, im N-terminalen Bereich (NTD) und an der Furin-Erkennungsregion (Furin-Cleavage Site), an der die beiden S-Protein-Domänen enzymatisch in den S1- und S2-Teil hydrolysiert werden.
Mutationen im Einzelnen
Eine Deletion an den Positionen H69 und V70 im Spike-Protein findet sich zusammen mit einigen anderen Mutationen auch in der Alpha-Variante. Sie verändert die Antigenität des S-Proteins und führt dazu, dass der S-Assay im PCR-Test ein falsch-negatives Ergebnis liefert:
Ein PCR-Target ist negativ, die anderen beiden positiv. Dieses S-Gen-Target-Failure (SGTF) genannte Phänomen liefert einen nützlichen Anhaltspunkt für die Prävalenz dieser Variante. So kann Omikron schon bei einem normalen PCR-Test detektiert werden, die Bestätigung erfolgt dann mittels Sequenzierung.