SARS-CoV-2 ist ein kugelförmiges Virion mit einem positivsträngigen viralen RNA-Genom, das in strukturelle und nicht-strukturelle Proteine translatiert wird. Die Größe des Genoms beträgt etwa 29 kbp, mit 14 offenen Leserahmen (ORFs) und 27 Proteinen, darunter vier Strukturproteine: das Spikeprotein (S), das Hüllprotein (E), das Membranprotein (M) und das Nukleokapsid (N).
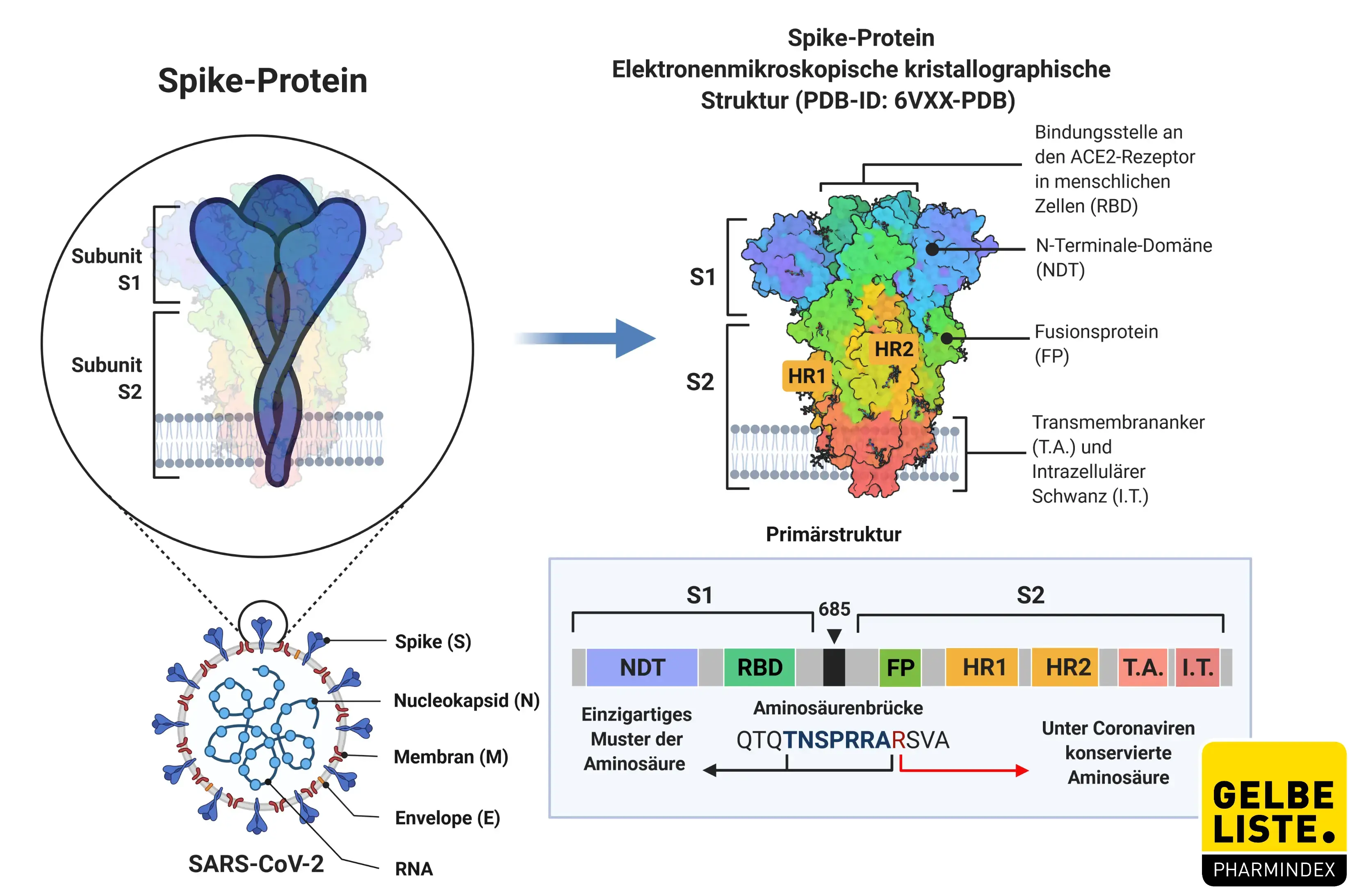
Das Spike-Protein ist ein Homotrimer bestehend aus 1273 Aminosäureresten, das sich auf der Oberfläche des Coronavirus befindet und eine entscheidende Rolle bei der Erkennung des Angiotensin-Converting-Enzym-2 (ACE-2) des Oberflächenrezeptors menschlicher Wirtszellen spielt. Für die Fusion von Virus- und Wirtszellen ist diese Erkennung notwendig, um den Transfer des viralen Nukleokapsids in die Wirtszellen zu initiieren.
Struktur
Das Spike-Protein umfasst eine N-terminale S1-Untereinheit und eine C-terminale membrannahe S2-Untereinheit. Die S1-Untereinheit besteht aus S1A-, S1B-, S1C- und S1D-Domänen.
- Die S1A-Domäne, die als N-terminale Domäne (NTD) bezeichnet wird, erkennt Kohlenhydrate, wie beispielsweise Sialinsäure, die für die Anheftung des Virus an die Wirtszelloberfläche erforderlich ist.
- Die S1B-Domäne, die Rezeptorbindungsdomäne (RBD) mit den Resten 319 bis 541 interagiert mit dem menschlichen ACE-2-Rezeptor. Die Strukturelemente innerhalb der S2-Untereinheit umfassen drei lange α-Helices, mehrere α-helikale Segmente, verlängerte verdrillte β-Faltblätter, membranüberspannende α-Helix und ein intrazelluläres Cystein-reiches Segment. Die Spike-RBD ist das Hauptziel neutralisierender Antikörper.
Das zwischen den S1- und S2-Untereinheiten in SARS-CoV-2 lokalisierte PRRA-Sequenzmotiv präsentiert eine Furin-Spaltungsstelle. In der S2-Untereinheit befindet sich eine zweite proteolytische Spaltungsstelle S2′, stromaufwärts des Fusionspeptids. Diese beiden Schnittstellen nehmen am viralen Eintritt in die Wirtszellen teil.
Mutationen, welche die Übertragbarkeit und die Pathogenität erhöhen
Die Mutationsrate bei SARS-CoV-2 beträgt etwa 8 × 10−4 Nukleotide/Genom jährlich, was für RNA-Viren sehr hoch ist. Coronaviren enthalten ein Exonuklease-Enzym, das ihre Replikationsfehlerrate in vitro um etwa das 15- bis 20-Fache reduziert, was zu einer etwa 10-fach niedrigeren viralen Mutationsrate in vivo führt als die von Influenza. Die Evolutionsrate von SARS-CoV-2 von Dezember 2019 bis Oktober 2020 ergab, dass das Virus in der Weltbevölkerung ungefähr zwei Mutationen pro Monat erwarb.
Die Hauptsorge im Zusammenhang mit den Varianten besteht in der potenziellen Beeinträchtigung der Antikörperneutralisation und der Wirksamkeit von Impfstoffen. Varianten, die sich weit verbreitet haben und Anzeichen dafür zeigten, dass sie leichter übertragbar sind, schwerere Krankheitsverläufe verursachen und/oder die Neutralisation durch Antikörper verringern, wurden von der Weltgesundheitsorganisation (WHO) als besorgniserregende Varianten (VOCs) eingestuft.
Der erste Hinweis auf einen genetischen evolutionären Selektionsdruck von SARS-CoV-2 wurde offensichtlich, als Anfang 2020 eine neue Virusvariante mit der Spike-Mutation D614G auftauchte und bis Juni 2020 auf eine Prävalenz von fast 100 Prozent anstieg.
Nach dem Auftauchen der Mutation D614G wurde in Schottland im März 2020 eine zunehmende Häufigkeit einer Aminosäuresubstitution, N439K, innerhalb des Rezeptorbindungsmotivs festgestellt. Während diese erste Linie mit N439K (bezeichnet als B.1.141) schnell verdrängt wurde, verbreitete sich parallel eine weitere Abstammungslinie (B.1.258), die N439K unabhängig erworben hatte. Bis Ende 2020 und Anfang 2021 wurden auch mehrere Varianten mit wiederkehrenden Mutationen (neben D614G) berichtet, die hauptsächlich, aber nicht ausschließlich, im Spike-Protein vorkommen.
N439K führt dazu, dass die Bindungsaffinität für den ACE2-Rezeptor erhöht und die neutralisierende Aktivität einiger monoklonaler und polyklonaler Antikörper reduziert wird. Eine weitere RBD-Aminosäuremutation, Y453F, die mit einer erhöhten ACE2-Bindungsaffinität verbunden ist, erhielt nach ihrer Identifizierung beträchtliche Aufmerksamkeit; insbesondere eine in Dänemark identifizierte Abstammungslinie, die ursprünglich als „Cluster 5“ (jetzt B.1.1.298) bezeichnet wurde.
Die B.1.1.298-Linie weist zudem Δ69–70 auf, eine Deletion der aminoterminalen Domäne (NTD), welche die RT-PCR-Testung teilweise stört und die virale Infektiosität stört sowie bei immungeschwächten Patienten zu einer Immunevasion führt.
Die Deletion Y144/145del verändert offenbar die Form des Spike-Proteins so, dass es einigen Antikörpern entkommen kann.
Die Mutation P681H befindet sich unmittelbar neben der Furin-Spaltungsstelle, was auf eine mögliche Rolle bei der Spike-Protein-Verarbeitung hindeutet.
Die E484K-Mutation (Vorkommen in Beta und Gamma) scheint einen Einfluss auf die Immunantwort und möglicherweise auf die Wirksamkeit der Impfstoffe zu haben, da sie mit einer erhöhten Bindung an menschliches ACE2 verbunden ist und dafür verantwortlich sein könnte, dass das Virus nicht mehr richtig von Antikörpern erkannt wird.
N501Y (Vorkommen in Alpha, Beta und Gamma) erhöht die Affinität des Spike-Proteins zu ACE2 durch Wasserstoffbrückenbindung mit ACE2 Y41 und erhöht die Virusreplikation in menschlichen Zellen der oberen Atemwege. N501Y beeinflusst jedoch nicht die Bindung und Neutralisation der meisten monoklonalen Antikörper.
K417N (Vorkommen in Beta und Gamma) trägt nicht zur ACE2-Bindung bei, ist jedoch ebenso wie E484K ein Schlüsselepitop für die Bindung neutralisierender Antikörper, wodurch die humorale Reaktion umgangen werden kann.
L452R (Vorkommen in Delta): Für den isolierten L452R Aminosäureaustausch wurde in vitro gezeigt, dass mutierte Viruspartikel höhere ACE2-Rezeptor-Affinität und verstärkte Infektiosität aufweisen. Laborexperimentelle Daten weisen außerdem darauf hin, dass diese Mutation eine Veränderung der antigenen Eigenschaften mit sich bringt
Die Alpha- und Delta-Varianten sind jeweils mit einer erhöhten Übertragbarkeit und einem größeren COVID-19-Schweregrad aufgrund von Immunevasion und möglicherweise aufgrund höherer Virusspiegel, die aus dem Antagonismus der angeborenen Immunität resultieren, verbunden. Die Beta- und Gamma-Varianten sind aufgrund ihrer Fähigkeit die humorale Immunität zu umgehen und Reinfektionen zu verursachen, jeweils mit einer erhöhten Übertragbarkeit verbunden.